|
Inhaltsübersicht | Nanomaschinen | Moleküle | Programme | Kurse | Fun | Links |
||
| > |
Menübalken der Jmol Applikation
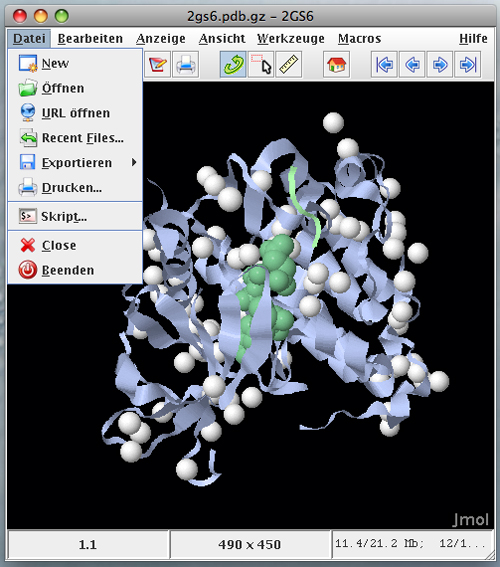
Grundfunktionen wie das Oeffnen von Koordinaten-Dateien und Einlesen von externen Skripten, exportieren von Bildern und einfache Darstellungsoptionen können hier über den Menübalken am oberen Rand des Applikationsfensters aufgerufen werden (oben rechts).
Im "Datei"-Menü kann mit "Oeffnen" ein lokales File geöffnet werden.
Mit "URL öffnen" kann man direkt ein Koordinatenfile aus einer Datenbank im Internet laden
(zB. "http://www.rcsb.org/pdb/files/2gs6.pdb.gz", um die Struktur 2gs6 der PDB-Datenbank zu laden).
"Exportieren" erlaubt das Abspeichern von Bildern in verschiedenen Formaten (.jpg, .png, .ppm), sowie den Export der Darstellung als .pdf-Datei, Zudem kann man die 3D-Szene an das ebefalls kostenlos erhältliche Ray-Tracing-Programm Pov-Ray weitergeben, das eine hochaufgelöste Darstellung erzeugt.
Mit "Export to Web Page" lässt sich direkt eine einfache Webseite erzeugen, die die in der Applikation aufgebaute Ansicht des Moleküls enthält.
Diese Optionen sind nur in der Applikation zugänglich, da das Web-Applet nicht auf lokale Dateien zugreifen kann.
Die Optionen der anderen Menüs sind auch über das Kontextmenü und über die Skriptkonsole aufrufbar, und werden im Kontektmenü besprochen.